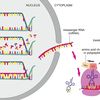
Bagaimana Cara Mengetahui Struktur Protein yang Belum Diketahui?
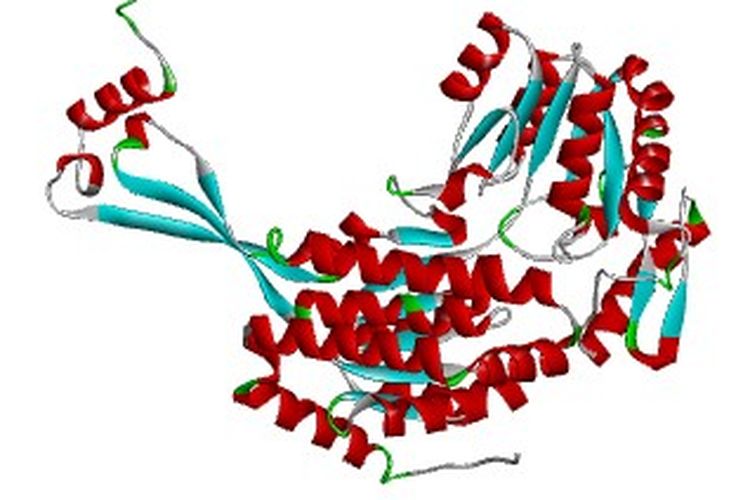
Biasanya ab initio menghasilkan sejumlah kemungkinan konformasi atau structure decoys.
Walau ab initio dapat dijadikan alternatif dalam pemodelan struktur 3D protein, namun metode ini memiliki akurasi yang rendah dan terbatas pada protein kecil dengan panjang asam amino kurang dari 120 residu.
Threading modeling menggunakan strategi penyelarasan struktur dan pelipatan sekuens. Metode ini digunakan ketika kemiripan sekuens asam amino target dengan template kurang dari 50 persen, atau dibawah kisaran metode homology modeling.
Jika pemodelan tetap dilakukan menggunakan homology modeling, beberapa asam amino tidak dapat dimodelkan karena tidak ada template yang sesuai.
Metode threading modeling memprediksi struktur 3D protein target, dengan menyelaraskan sekuens ke struktur eksperimental serupa dengan yang ada pada Protein Data Bank (PDB).
Dalam metode ini, ketika dua protein memiliki sequence identity kurang dari 25 persen, dapat dipastikan akurasi keselarasannya akan sangat rendah.
Dikenal dengan pemodelan komparatif. Adalah pemodelan yang membangun struktur protein 3D yang belum diketahui, dengan membandingkan dan memanfaatkan ketersediaan informasi dari sekuens protein homolog.
Metode ini digunakan ketika kemiripan sekuens asam amino target dengan template lebih besar dari atau sama dengan 50 persen.
Baca juga: Definisi dan Struktur Protein Kuartener
Homology modeling memiliki akurasi yang tinggi pada kesamaan sekuens, dengan tingkat kesalahan cukup rendah dalam memodelkan rantai samping maupun penentuan posisi loop.
Struktur 3D yang dibentuk melalui homology modeling memiliki kemiripan yang baik dengan struktur yang biasa diselesaikan dengan eksperimen laboratorium menggunakan Nuclear Magnetic Resonance (NMR).
Aplikasi untuk memprediksi struktur protein
Jika dilakukan lewat eksperimen laboratorium, aplikasi prediksi struktur protein tidak diperlukan seperti pada eksperimen in silico.
Sebagai tambahan informasi, berikut beberapa contoh aplikasi untuk memprediksi struktur protein secara in silico:
QUARK
Merupakan salah satu aplikasi yang digunakan pada metode prediksi struktur ab initio. Aplikasi ini dapat digunakan secara online dan bebas biaya.
Aplikasi QUARK dapat diakses melalui QUARK.
I-TASSER
Merupakan salah satu aplikasi yang digunakan pada metode prediksi struktur threading modeling. Aplikasi ini juga dapat digunakan secara online dan bebas biaya.
Aplikasi I-TASSER dapat diakses melalui I-TASSER.
SWISS-MODEL
Merupakan salah satu aplikasi yang digunakan pada metode prediksi struktur homology modeling. Aplikasi ini juga dapat digunakan secara online dan bebas biaya.
SWISS-MODEL dapat diakses melalui SWISS-MODEL.
Baca juga: Proses Metabolisme Protein: Bagaimana Tubuh Mencerna Protein?
Simak breaking news dan berita pilihan kami langsung di ponselmu. Pilih saluran andalanmu akses berita Kompas.com WhatsApp Channel : https://www.whatsapp.com/channel/0029VaFPbedBPzjZrk13HO3D. Pastikan kamu sudah install aplikasi WhatsApp ya.